孙育杰课题组超分辨成像解析Cohesin复合体在三维基因组构建中的分子机制
信息来源:孙育杰教授团队
哺乳动物细胞间期核内基因组呈现高度有序的层级结构。2m长的DNA经过不断压缩折叠储存在细胞核中并依次形成不同尺度的结构域:核小体、染色质纤维、染色质环、拓扑相关结构域(topologically associating domains, TADs)、区室(compartments)和染色体领地(chromosome territory),其中每一个层级结构都蕴含了丰富的DNA互作网络,共同实现细胞生命历程中的调控。基因组层级结构的形成与维持受到多种蛋白因子的调控,如架构蛋白、转录因子和非编码RNA等。其中部分染色质环的形成依赖于CTCF、cohesin和装载复合物NIPBL-MAU2等架构蛋白,cohesin通过“环挤出(Loopextrusion)”的方式使染色质环逐渐增大,最终形成较为稳定的局部互作结构域,而CTCF和cohesin结合在其基部。然而,cohesin和NIPBL 协同促进染色质挤出的具体机制尚不明确。
2023年6月28日,北京大学未来技术学院国家生物医学成像科学中心(NBIC),生物医学前沿创新中心(BIOPIC),膜生物学国家重点实验室孙育杰课题组发表了题为“Rad21 is the core subunit of the cohesin complex involved in directing genome organization”的研究论文(图1),在这项研究中,作者使用超高分辨率显微镜成像技术揭示了cohesin复合物的RAD21亚基在cohesin装载和染色质结构调控中的独特作用。

图1 论文截图
本研究中,作者首先观察到RAD21的过表达导致染色质环被过度挤出,在细胞核中形成“意大利面”样的分布,而RAD21蛋白自身会聚集成簇(图2)。进一步研究发现,过度装载在染色质上的cohesin蛋白会形成“蝴蝶结”样结构并绑住TAD(图3),最终形成“念珠”状分布,而其他四种cohesin亚基的过表达却在细胞核内呈现均匀分布。
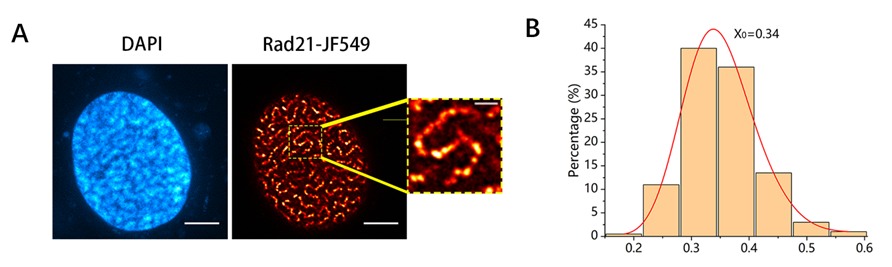
图2 在HeLa细胞中过表达RAD21亚基后在细胞核内形成的“意大利面”样结构
利用RAD21与装载复合物NIPBL-MAU2的互作突变体RAD21-LIS,生化实验及细胞成像实验证实了该互作的重要性,这种互作促进了cohesin在染色质上的大量装载,最终形成“意大利面”样分布。
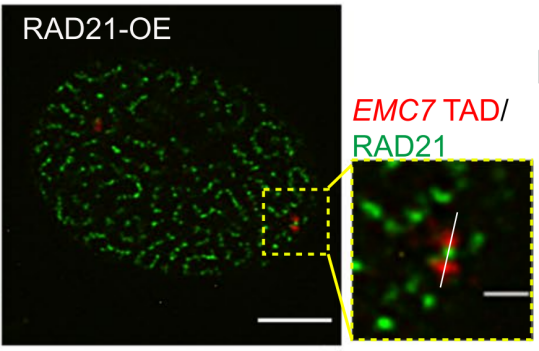
图3 RAD21过表达后RAD21和EMC7TAD的超高分辨显微成像
此外,本文通过分析Hi-C数据,对比了RAD21上调后和先前研究报道的同样能够造成染色质“意大利面”样分布的WAPL蛋白缺失对染色质不同层级结构(全基因组范围、染色质区室和TADs)的影响异同,以进一步解释RAD21在染色质结构方面的作用机制(图4)(Haarhuis et al., 2017; Wutz et al., 2017)。作者发现“意大利面”样分布形成后,在TAD的边角处积累了大量的互作信号,同时TAD之间的相互作用明显增加。但是,RAD21的上调对TADs的长度和数目并没有显著影响,并且TAD之间的边界的绝缘程度也没有显著改变,这一现象与WAPL缺失带来的结果不同。作者推测可能是RAD21的上调使得不同TADs被更加稳定地分隔,因此TADs长度、数目以及绝缘系数并未受到显著影响。这可能是其作用机制区别与WAPL,有待于进一步研究。
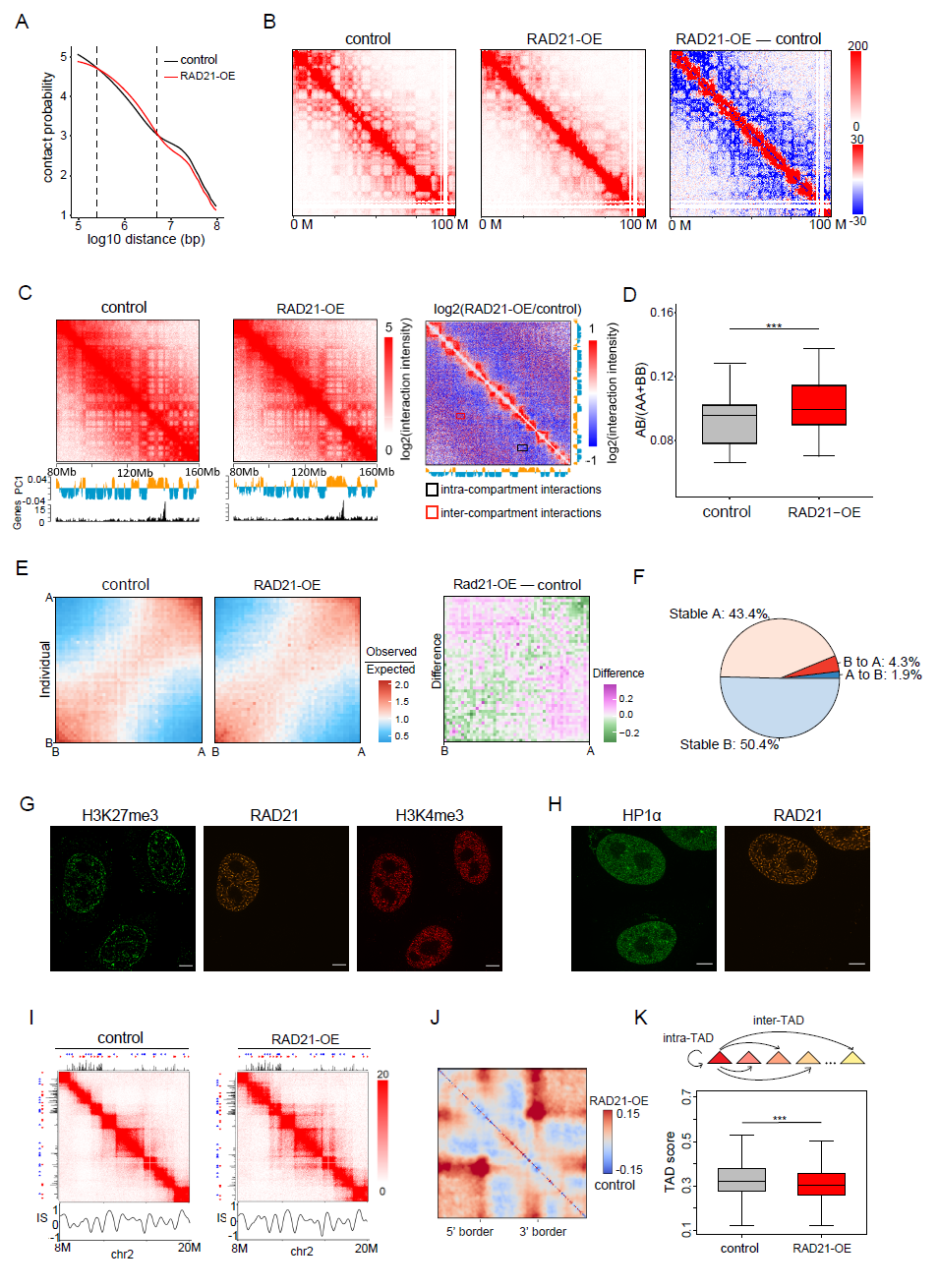
图4 Hi-C数据分析RAD21上调对染色质不同层级结构的影响
最后,结合临床数据和细胞实验,作者发现在乳腺癌病例中患者生存率的降低与RAD21的异常表达十分相关,而在乳腺癌细胞系中的免疫荧光成像表明,内源的RAD21呈现出与过表达RAD21的HeLa细胞中类似的簇状分布。结合RNA-seq和Hi-C分析,作者发现HeLa细胞中RAD21上调表达后会导致染色质compartment切换及癌症相关基因的显著上调,而这种基因组结构的异常可能会导致乳腺癌肿瘤的发生。
综上所述,本研究深入研究了RAD21亚基促进cohesin装载过程的分子机制,同时提出了cohesin和装载复合物如何协同配合并促进染色质挤出的模型(图5),该模型对细胞内部三维基因组结构的构建具有重要的意义,此外本研究描述染色质状态与癌症病因之间的重要关系,为临床治疗的靶点信息提供了重要参考。
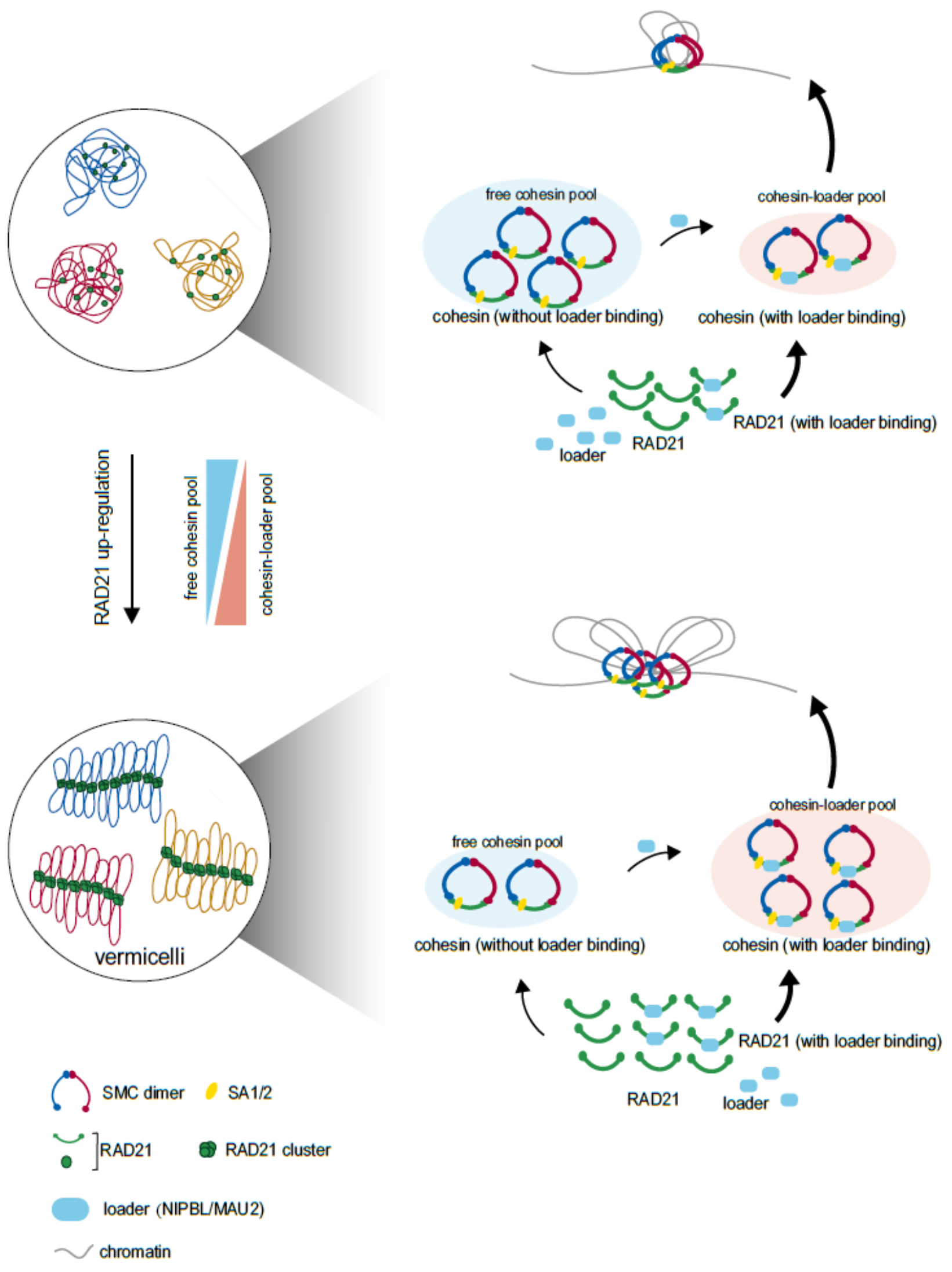
图5 RAD21作为cohesin的核心亚基帮助细胞内“意大利面”样分布形成的模型
北京大学生命科学学院已毕业博士生孙雨傲、博士生许鑫和博士生赵文雪为本文的并列第一作者,孙育杰教授为通讯作者。实验室成员陈珂旸、李永正、王啸天、张梦凌、薛博鑫、余婉婷对文章也做出了重要贡献。北京大学人民医院王殊教授及王朝斌博士在乳腺癌组织及细胞方面做出了重要合作,北京大学生命科学学院孔道春教授在本研究的生化实验中提供了重要支持,北京大学生命科学学院李程教授和清华大学生命科学学院颉伟教授及实验室成员张宇博士在测序数据的分析方面提供了重要帮助。本研究得到了国家重点研发计划(编号:2022YFA1303103)和国家基金委杰出青年基金(编号:21825401)的资助,以及北京大学生命科学学院仪器中心及凤凰工程平台的技术支持。
文章链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-023-02982-1
参考文献:
1. Haarhuis, J.H.I., van der Weide, R.H., Blomen, V.A., Yanez-Cuna, J.O., Amendola, M., van Ruiten, M.S., Krijger, P.H.L., Teunissen, H., Medema, R.H., van Steensel, B., et al. (2017). The Cohesin Release Factor WAPL Restricts Chromatin Loop Extension. Cell 169, 693-707.
2. Wutz, G., Varnai, C., Nagasaka, K., Cisneros, D.A., Stocsits, R.R., Tang, W., Schoenfelder, S., Jessberger, G., Muhar, M., Hossain, M.J., et al. (2017). Topologically associating domains and chromatin loops depend on cohesin and are regulated by CTCF, WAPL, and PDS5 proteins. EMBO J 36, 3573-3599.




