Nature Methods | 何爱彬团队开发单细胞药物基因组靶向检测新技术解析表观耐药机制
信息来源:何爱彬教授团队
小分子药物与基因组的结合构成了广泛的疾病治疗的分子基础,包括传统药物和靶向治疗。尽管大量的小分子药物通过与基因组互作的效果在临床治疗中得到了充分验证,但许多的小分子具体机制仍不清楚,耐药产生机制也尚未可知。绘制靶向基因组或染色质相关蛋白的药物多组学图谱,对于解读它们在抗癌治疗中的功能与耐药复发机制至关重要。迄今为止,只有少数技术能够在大量细胞群体(bulk)水平测量小分子与细胞基因组的互作【1-3】,单细胞多组学水平解析小分子与基因组表观组互作尚未实现。因此,亟待开发高通量的、普适性的药物基因组结合的单细胞检测技术。

2024年7月18日,北京大学未来技术学院/国家生物医学成像科学中心何爱彬团队与合作者在《Nature Methods》杂志在线发表题为“Single-cell EpiChem jointly measures drug-chromatin binding and multimodal epigenome”的研究论文,报道了一种全新高通量的名为scEpiChem(Single Cell co-assay of Epigenome and small molecule Chemicals)的单细胞药物多组学技术,这个方法能够同时在单细胞中捕获基因组药物靶标互作和多模态表观遗传状态,解析药物反应和功能异质性以及耐药分子机制。
何爱彬团队长期致力于单细胞表观多组学底层新技术研发,目前已建立一系列普适性、高精度的单细胞ChIP-seq技术(scitChIP、CoBATCH)与单细胞表观多组学技术(CoTECH、uCoTargetX)【4-7】。scEpiChem通过使用多轮的组合标签(spilt &pool)和抗体-PAT T7 complex顺序切割的策略,实现了超高通量、简单易用的药物多组学检测技术,可以同时捕获百万个单细胞的基因组药物-靶点的结合和多模态表观遗传图谱(图1)。
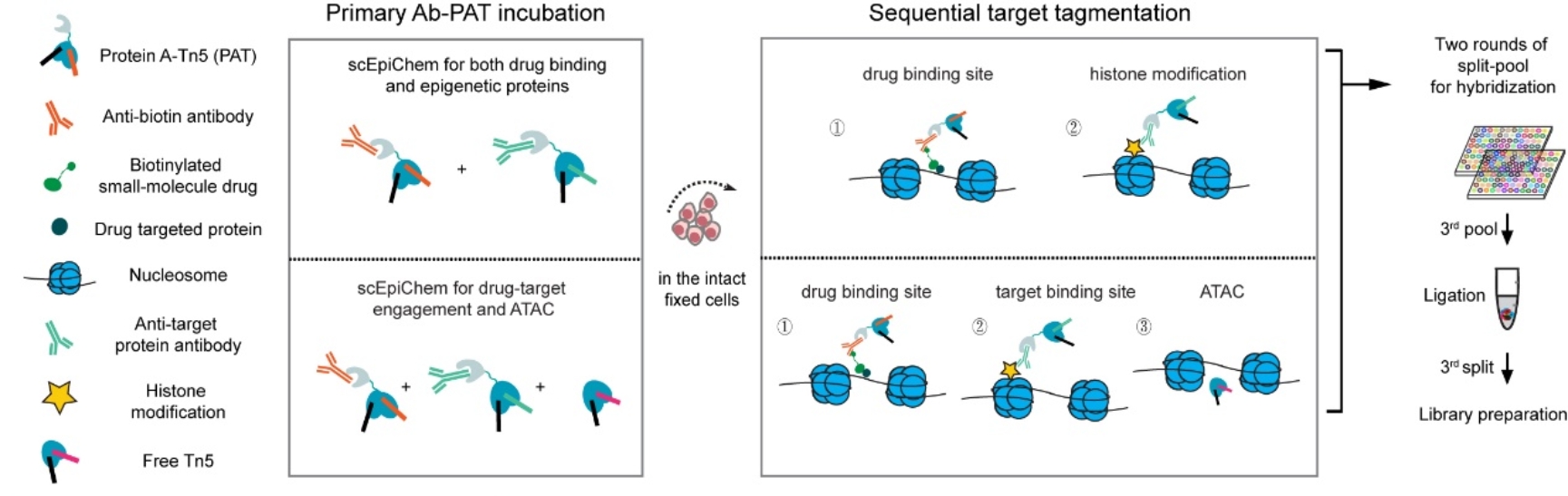
图1. 高通量单细胞药物多组学技术scEpiChem流程图
首先,研究者利用scEpiChem在细胞系上对BET溴结构域抑制剂JQ1和CDK7的抑制剂THZ1,以及其他表观基因组特征进行了同时分析。结果表明,scEpiChem能够准确识别各细胞类型中小分子药物-靶点结合,组蛋白修饰或者是染色质可及性特征(图2)。
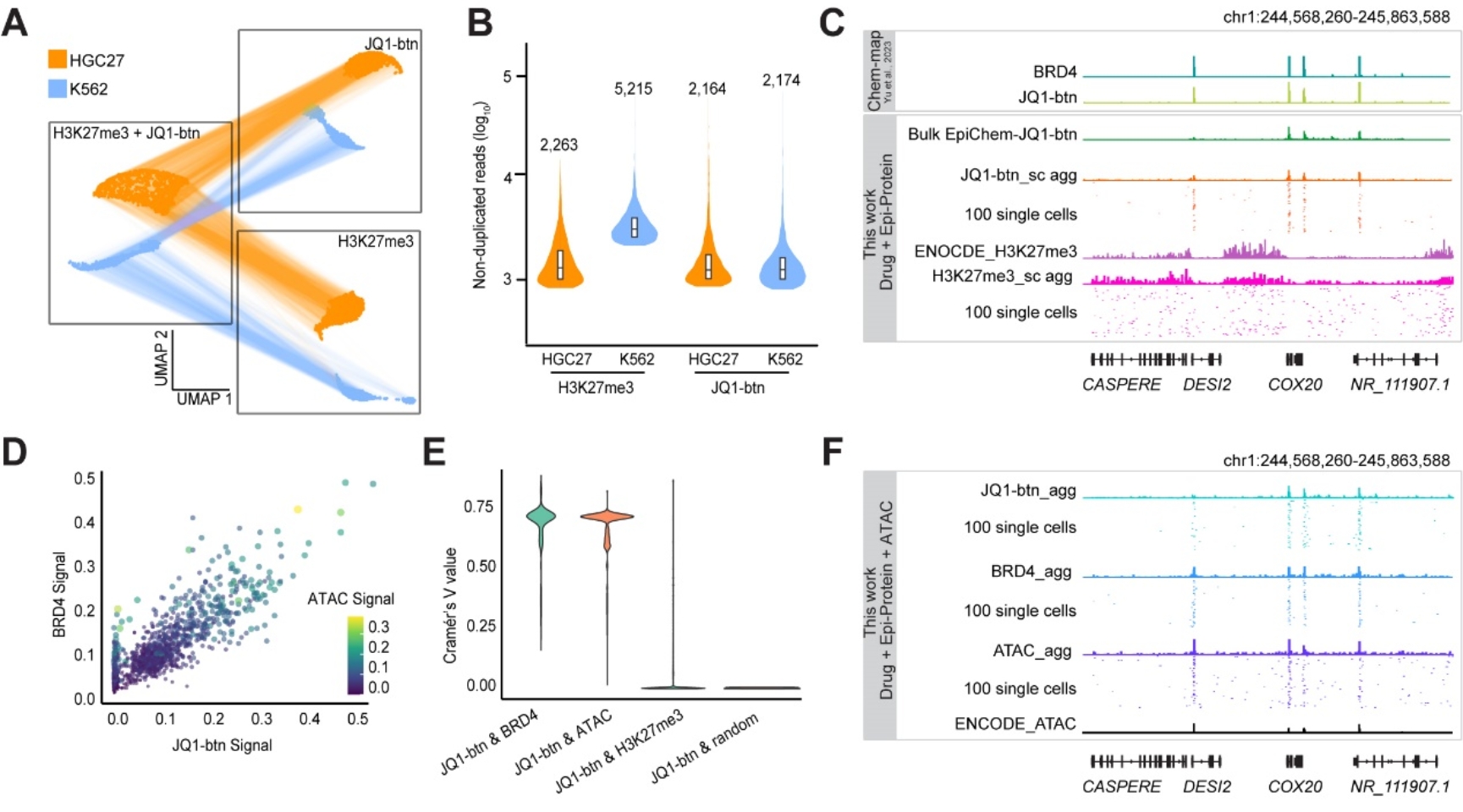
图2. scEpiChem实现基因组药物-靶点结合和多模态表观分析策略
scEpiChem提供了一个全面的高通量分析框架,捕捉了传统bulk分析无法实现的小分子药物反应的细胞异质性特征。例如,经历上皮-间质转化(EMT)的结直肠癌(CRC)类器官显示出不同细胞类型对药物治疗反应的变化,识别出各细胞类型药物-染色质结合与靶蛋白-染色质结合之间的细微差异,从而提供了药物与其已知蛋白质靶点共享和独特结合位点的见解(图3)。在药物耐药性研究中,通过同时映射单细胞中的小分子药物和组蛋白修饰特征,scEpiChem为研究药物与动态表观基因组景观之间的多样化相互作用开辟了新途径,识别出一系列可能参与药物反应的转录因子。这些知识对于设计更有针对性和更有效的治疗策略至关重要,例如组合选定的小分子药物以克服耐药性【1】。
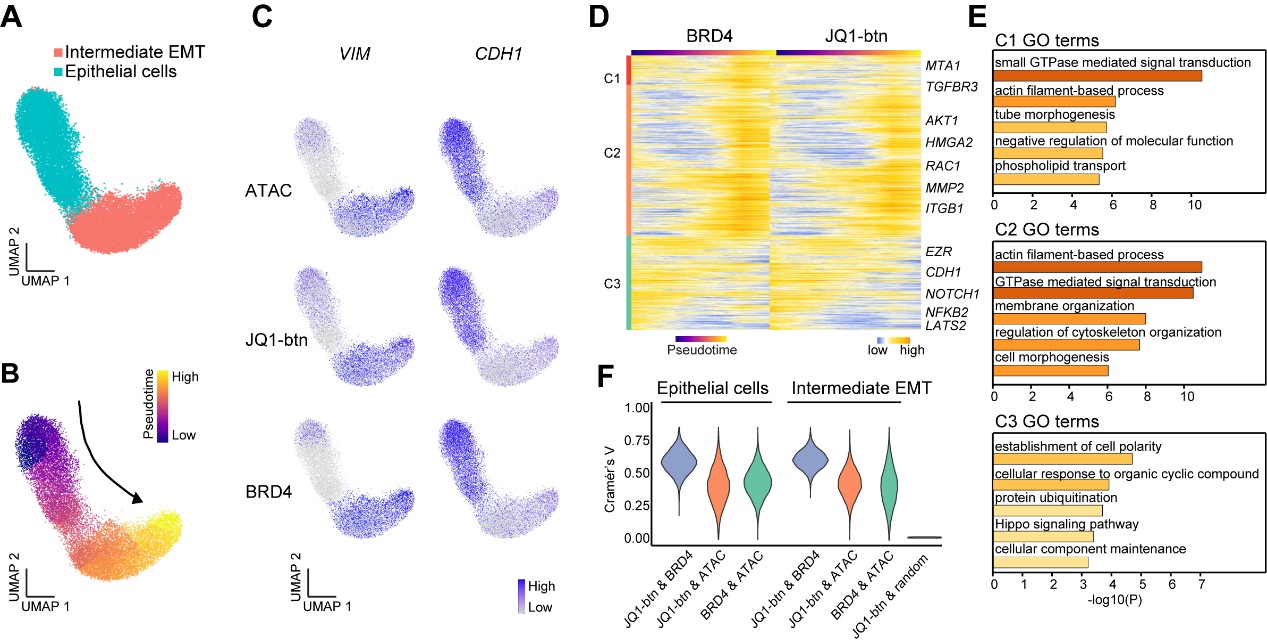
图3. scEpiChem揭示肿瘤微环境药物反应和功能异质性
北京大学未来技术学院博士生董超和张彤、北京大学-清华大学生命科学联合中心博士生孟晓萱以及北京大学肿瘤医院博士后郭志芳为论文共同第一作者。何爱彬教授和舒绍坤研究员为本文的共同通讯作者。中国医学科学院血液学研究所血液病医院熊海清研究员在研究中有重要贡献。该研究获得了科技部重点研发项目、国家自然科学基金委、北京大学成都前沿交叉生物技术研究院和生命科学联合中心的支持。
原文链接:DOI: 10.1038/s41592-024-02360-0
参考文献:
1. Anders, L. et al. Genome-wide localization of small molecules. Nature Biotechnology32, 92-96 (2014).
2. Tyler, D.S. et al. Click chemistry enables preclinical evaluation of targeted epigenetic therapies. Science 356, 1397-1401 (2017).
3. Yu, Z. et al. Chem-map profiles drug binding to chromatin in cells. Nature Biotechnology (2023).
4. Wang, Q. et al.CoBATCH for High-Throughput Single-Cell Epigenomic Profiling. Mol Cell76, 206-216 e207 (2019).
5. Ai, S. et al. Profiling chromatin states using single-cell itChIP-seq. Nat Cell Biol21, 1164-1172 (2019).
6. Xiong, H. et al. Single-cell joint detection of chromatin occupancy and transcriptome enables higher-dimensional epigenomic reconstructions. Nat Methods18, 652-660 (2021).
7. Xiong, H. et al. Single-cell joint profiling of multiple epigenetic proteins and gene transcription. Sci Adv10, eadi3664 (2024).




